使用可解释的AI进行结肠直肠癌分类的个性化肠道微生物组分析
肠道微生物群包含对人类健康至关重要的不同细菌种类的复杂种群。近年来,多个领域的科学家发现,肠道微生物组的变化可能与多种疾病有关,尤其是结直肠癌(CRC)。多项研究表明,某些细菌的丰度较高,例如具核梭杆菌和微单胞菌,通常与 CRC 进展有关。
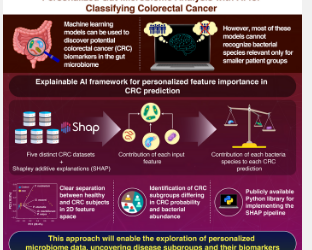
基于这些发现,研究人员开发了各种人工智能 (AI) 模型来帮助他们分析哪些细菌种类可用作 CRC 生物标志物。然而,这些模型中的大多数都依赖于所谓的“全局解释”,这意味着它们只能考虑整个输入数据来做出预测。因此,此类模型无法识别可能作为较小、代表性较低的患者群体的相关 CRC 生物标志物的细菌种类。
在此背景下,东京工业大学 (Tokyo Tech) 的一个研究团队决定采用一种能够解决这一局限性的不同方法。正如他们最近发表在Genome Biology上的论文中所述,该团队采用了一个可解释的 AI 框架,该框架为其 CRC 预测提供本地而非全球解释。该研究的主要作者山田拓司副教授解释说:“局部解释技术使我们有可能发现对每个 CRC 患者影响最大的细菌,从而使我们能够检查疾病组内受试者之间的个体差异。”
该团队使用了一个名为“Shapley additive explanations”(SHAP)的框架,该框架源自博弈论中称为 Shapley 值的概念。简而言之,Shapley 值告诉我们应该如何在联盟或团体的玩家之间分配支出。同样,在他们的研究中,该团队使用 SHAP 来计算每个细菌物种对每个单独的 CRC 预测的贡献。
使用这种方法以及来自五个 CRC 数据集的数据,研究人员发现将 SHAP 值投影到二维 (2D) 空间中可以让他们看到健康受试者和 CRC 受试者之间的明显区别。将此 2D 信息聚类产生四个不同的 CRC 受试者亚组,每个亚组的 CRC 概率和相关细菌都不同。此外,该团队发现,CRC 概率最高的 CRC 亚组中的受试者总是拥有丰富的通常与 CRC 相关的细菌群。最值得注意的是,五个数据集的结果是一致的,展示了该方法的广泛适用性。
有了这些有希望的结果,该团队预计他们的方法将为肠道微生物组研究界做出坚实的贡献。“考虑到机器学习在微生物组-疾病关联研究中的使用越来越多,我们的新方法可能有利于更个性化的微生物组数据探索,并有助于发现潜在的疾病亚组及其潜在的相关生物标志物,”Yamada 博士推测。此外,该技术还适用于已知与肠道微生物组相关的其他疾病,例如溃疡性结肠炎、克罗恩氏病和糖尿病。
希望可解释的人工智能在不久的将来能揭示更多肠道微生物组的秘密,敬请期待!
免责声明:本文由用户上传,与本网站立场无关。财经信息仅供读者参考,并不构成投资建议。投资者据此操作,风险自担。 如有侵权请联系删除!
