微观和宏观方法的结合为不同大脑区域的连接方式提供了新的思路
要了解我们的大脑是如何工作的,没有办法绕过研究不同的大脑区域是如何通过神经纤维相互连接的。在最新一期的《科学》杂志上,人类大脑计划 (HBP) 的研究人员回顾了该领域的现状,提供了有关大脑连接组如何在不同空间尺度上(从分子和细胞到宏观水平)构建的见解,以及评估现有方法和未来要求以了解连接组的复杂组织。
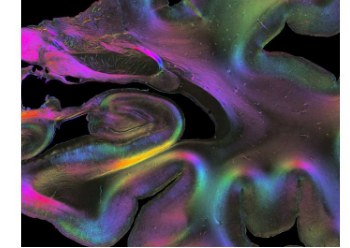
“仅用一种或两种方法研究大脑连接是不够的,”作者兼 HBP 科学主任 Katrin Amunts 说,他是 Forschungszentrum Jülich 和 C. & O 神经科学与医学研究所 (INM-1) 的负责人。杜塞尔多夫大学医院沃格特脑研究所。
“连接组嵌套在多个层次上。要了解它的结构,我们需要通过在多尺度方法中结合不同的实验方法并将获得的数据整合到多层次的地图集中,例如 Julich Brain Atlas,同时查看多个空间尺度我们已经开发出来的。”
来自 Forschungszentrum Jülich 和伍珀塔尔大学物理系的 Markus Axer 和他在 INM-1 的团队开发了一种独特的方法,称为 3D 偏振光成像 (3D-PLI) 来可视化神经显微分辨率的纤维。研究人员在连续的大脑切片中追踪纤维的 3D 过程,目的是开发整个人脑的 3D 纤维图谱。
与来自法国 Neurospin 和意大利佛罗伦萨大学的其他 HBP 研究人员一起,Axer 和他的团队最近使用几种不同的方法对人类海马的相同组织块进行了成像:解剖和扩散磁共振成像(aMRI 和 dMRI),两种-光子荧光显微镜(TPFM)和3D-PLI,分别。
像 TPFM 这样的显微镜方法提供了小脑容量的亚微米分辨率图像,揭示了大脑大脑皮层的微观结构,但它们在解开连接遥远大脑区域的纤维方面存在局限性,这些纤维构建了深部白质结构。对于电子显微镜测量来说更是如此,它能够以纳米分辨率洞察一立方毫米的脑组织。相比之下,dMRI 可用于全脑水平的纤维束成像——可视化白质连接——但不能分辨单个纤维或小束。
“3D-PLI 是微观和宏观方法之间的桥梁,”Amunts 说。“这是因为 3D-PLI 以高分辨率解析纤维结构,同时允许对全脑部分进行成像,然后我们可以在 3D 中重建以追踪纤维连接。”
结合 dMRI、TPFM 和 3D-PLI 使研究人员能够在同一参考空间内叠加三种模式。“这种数据整合只有通过对同一个组织样本进行成像才能实现,”Axer 解释说。人类海马体块从德国到法国,再回到德国,最后到意大利,在得益于当地高度专业化设备的不同实验室中进行处理和成像。
然后,研究人员使用Julich Brain Atlas将他们的数据在空间上锚定在解剖参考空间中。三维地图集包含超过 250 个大脑区域的细胞结构图,是HBP 多层次人脑地图集的核心。“我们的大脑图谱使我们能够准确地确定我们在大脑中发现这些微结构的位置,”Amunts 解释说。该数据集可通过 HBP 的 EBRAINS 基础设施公开访问,并可在交互式图集查看器中浏览。
研究人员的多尺度方法结合了不同空间尺度的多种模式来解开人类连接组是独一无二的,并为人类大脑的工作方式提供了令人兴奋的新见解。
尽管海马体重建是一个灯塔项目,但仍有几项国际努力正在进行(或即将开始),需要在开放的地图集层面进行协调,以实现多尺度数据的整合。Amunts 和 Axer 强调,这是在实验可访问的尺度范围内(从轴突到通路)揭示连通性原则的先决条件。
换句话说,需要一种结合微观和宏观方法的综合多尺度方法来描述和理解人脑的嵌套组织。作者说,这需要对当前的方法进行批判性的重新评估,包括纤维束成像。
免责声明:本文由用户上传,与本网站立场无关。财经信息仅供读者参考,并不构成投资建议。投资者据此操作,风险自担。 如有侵权请联系删除!
